R によるランドマーク解析: X 線と内視鏡検診による胃癌生存率
井口豊(生物科学研究所,長野県岡谷市)
最終更新: 2024 年 7 月 7 日
1. はじめに
医療論文を読んでいて,気になった生存解析があった。以下の論文である。
萩原廣明・茂木文孝・山下由起子・小板橋毅・八木茂・関口利和 (2016)
直接X線との対比による内視鏡胃がん検診の有用性
日本消化器がん検診学会雑誌 54(4): 518-527.
X 線と内視鏡診断による胃癌生存率を,カプラン・マイヤー(Kaplan-Meier)分析したグラフが, p.523 の図 8 と 9 に描かれている。
これは,ランドマーク解析(landmark analysis)しても良かったのではないかと考え, R を用いて改めて分析してみた。ここでは,とりあえず,図 8 のデータを分析した。
2. R によるランドマーク解析
ランドマーク解析には, R のパッケージ jskm の中の jskm 関数を利用した。これは,Kaplan-Meier 曲線を ggplot2 で描く。
生存率データは,図 8 グラフから読み取り,さらに,グラフ下に書かれた対象症例数の人数データも利用した。検診別のデータ(radiograph と endoscope)は, screening.dat としてダウンロードできる。カットオフ時点は,3 年とした。そのデータを含めたスクリプトは以下のとおりである。
# 入力データ
df<- read.table("screening.dat", header=T)
df # データフレーム確認
# 利用パッケージ
library(survival)
library(jskm)
### 生存率解析
fit<- survfit(Surv(time, event) ~ exam, data = df)
# 全体のカプラン・マイヤー曲線とログランク検定
jskm(
fit, cumhaz = F, mark = F,
xlims = c(0, 7),
surv.scale = "percent",
pval.testname = T, pval = T, table = T,
cut.landmark = NULL,
showpercent = T, timeby = 1, data = df,
legendposition = c(0.8, 0.2),
pval.coord = c(2, 0.2)
)
# ランドマーク解析(3 年カットオフ)
jskm(
fit, cumhaz = F, mark = F,
xlims = c(0, 7),
surv.scale = "percent",
pval.testname = T, pval = T, table = T,
cut.landmark = 3,
showpercent = T, timeby = 1, data = df,
legendposition = c(0.8, 0.4),
pval.coord = c(1.5, 0.2)
)
### 終了
3. 結果
まず全体の カプラン・マイヤー曲線とログランク(log-rank)検定で,以下のようなグラフで表される。
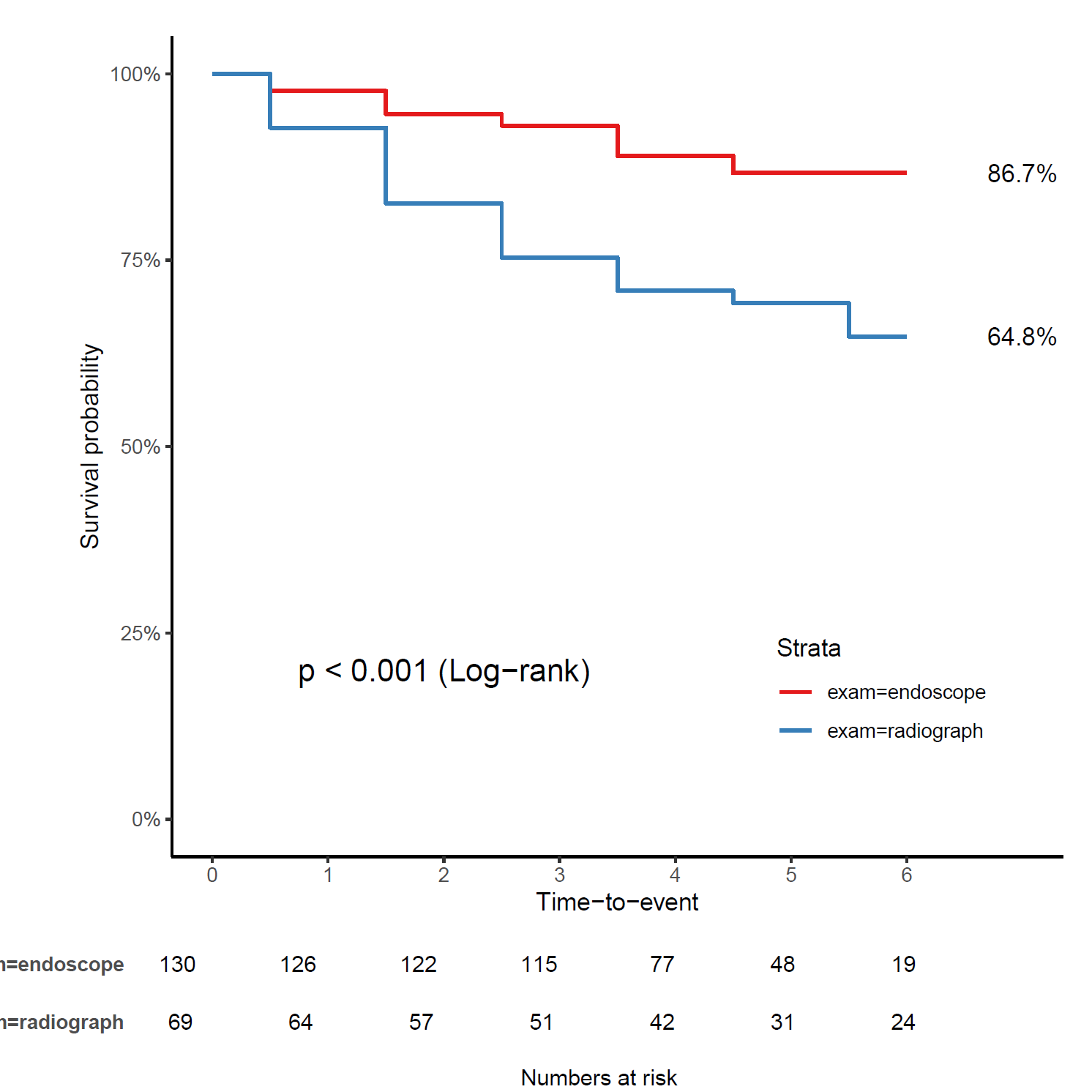
このデータの範囲内の最終的な生存率(5 年生存率に相当)が,論文では,内視鏡検診 86.8%,直接 X 線検診 68.4% であり,上記の再現データの分析では,内視鏡検診 86.7%,直接 X 線検診 64.8% であったので,元データをほぼ復元した分析だと言える。ログランク検定の結果も,どちらも 1% 水準で有意差あり,だった。
次に, 3 年でカットオフしたランドマーク解析の結果を図 2 に示す。
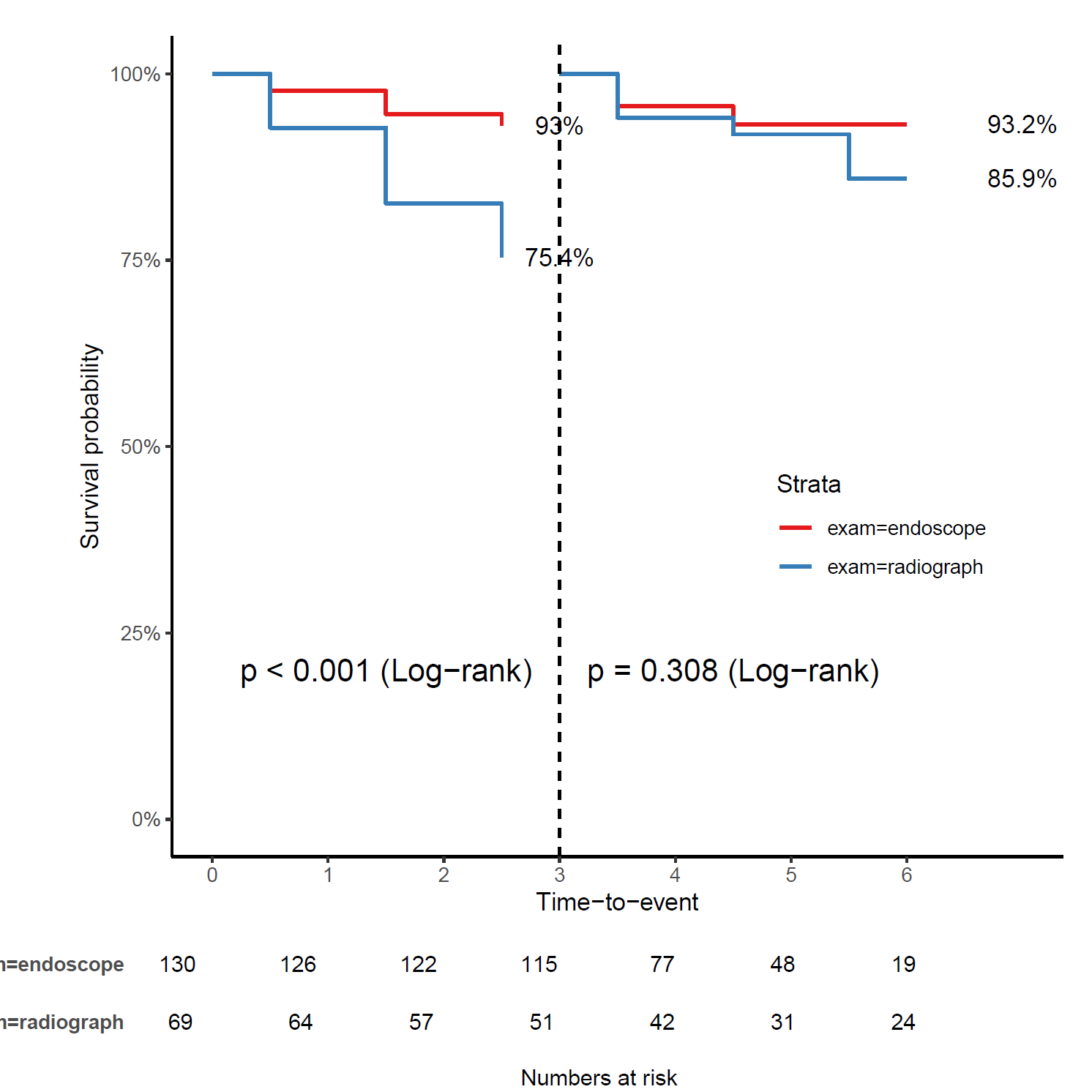
直接 X 線と内視鏡による胃がん検診で,生存率は, 3 年未満では 1% 水準で有意差が見られるが,3年以上では有意差が見られないことが分かる。
もちろん,生データを分析しないと正確なことは言えないが,ランドマーク解析もしてほしかった事例である。