対応ある Brunner-Munzel 検定: Wilcoxon符号順位検定の対称性条件の回避
井口豊(生物科学研究所,長野県岡谷市)
最終更新: 2025 年 3 月 2 日
PDF version
DOI: 10.5281/zenodo.14945777
1. はじめに
Brunner-Munzel 検定と言えば,一般的には,独立 2 群の検定,いわゆる,対応無いデータに対する検定として知られるが,対応有りの場合の検定もある。
対応有りのノンパラメトリック検定としては, Wilcoxon 符号順位検定が有名だが,これを中央値検定として利用するには,差データの分布対称という厄介な条件が付いてくる。
この条件を回避するために,対応ある Brunner-Munzel 検定が有用になる。
ここでは, R のパッケージ TOSTER の brunner_munzel 関数を利用した例を紹介する。
この関数の引数 paired を TRUE とするか, FALSE とするかで,対応ありなしを使い分ける。ヘルプの中で,重要だと思われる解説を抜き出しておく。
This tests the hypothesis that the relative effect, discussed below, is equal to the null value (default is mu = 0.5).
The estimate of the relative effect, which can be considered as value similar to the probability of superiority, refers to the following:
\begin{eqnarray}
\hat{p}=p(X < Y) + \frac{1}{2}P(X = Y)
\end{eqnarray}
Note, for paired samples, this does not refer to the probability of an increase/decrease in paired sample but rather the probability that a randomly sampled value of X.
この検定は,一般的に言われている,中央値の検定
,というよりも,相対効果(relative effect)の検定,と言うべきだろう。
2. 非対称な差データに対応ある Brunner-Munzel 検定
ここでは,前述のウェブサイト同様に,対数正規乱数データを使い,中央値 5 は同じで,分散が異なる対数正規母集団からの対応あるデータを取り出して, Wilcoxon 符号順位検定と対応ある Brunner-Munzel 検定のシミュレーションをおこなう。
#############
library(TOSTER)
n<- 100
set.seed(10)
k<- 1e+4
p<- replicate(k, {
x1 <- rlnorm(
n, meanlog = log(5), sdlog = 0.2
)
x2 <- rlnorm(
n, meanlog = log(5), sdlog = 0.8
)
c(
wilcox.test(x1, x2,
correct = F, paired = TRUE
)$p.value,
brunner_munzel(x1, x2,
paired = TRUE
)$p.value
)
})
library(Hmisc)
par(mfrow = c(2, 2))
# Wilcoxon p 値出現頻度ヒストグラム
hist(
p[1, ],
xlab = "p value",
freq = F,
main = "Wilcoxon signed-rank test"
)
# 対応ある Brunner-Munzel p 値出現頻度ヒストグラム
hist(
p[2, ],
xlab = "p value",
freq = F,
main = "Paired Brunner-Munzel test"
)
# 経験累積分布関数(ECDF)
ck<- 1:k/k
d.1<- c(ck, p[1, ])
d.2<- c(ck, p[2, ])
test.1<- relevel(
factor(
rep(c("Theoretical", "Wilcoxon signed-rank test"),
each = k)
),
ref = "Theoretical")
test.2<- relevel(
factor(
rep(c("Theoretical", "Paired Brunner-Munzel test"),
each = k)
),
ref = "Theoretical")
# Wilcoxon 符号順位和
Ecdf(
d.1,
xlab="p value",
label.curves=list(keys= 1:2),
lty = 1:2,
group = test.1,
main = "ECDF",
subtitles = FALSE
)
# 対応ある Brunner-Munzel
Ecdf(
d.2,
xlab="p value",
label.curves=list(keys= 1:2),
lty = 1:2,
group = test.2,
main = "ECDF",
subtitles = FALSE
)
################
結果は,次の図 1 のとおりである。
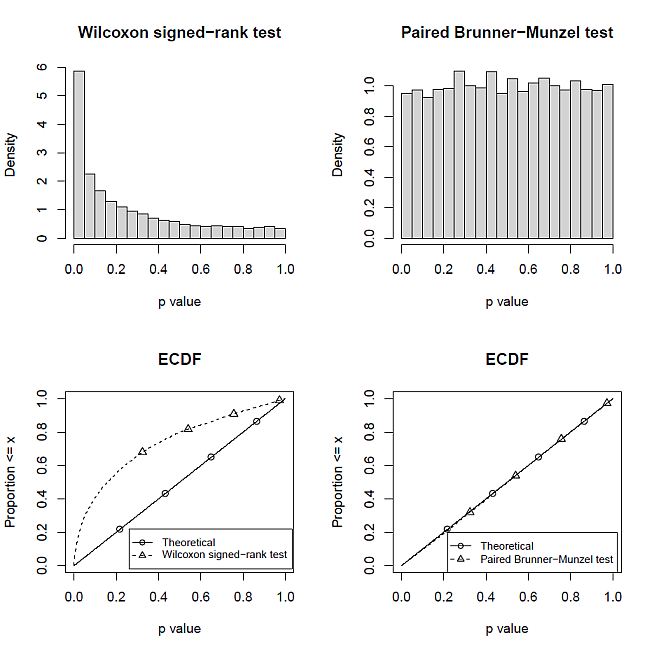
このデータを見る限りだが,やはり,対応ある Brunner-Munzel検定のほうが適切に検定できそうだ。
なお,この問題に関しては, X (旧 Twitter) の黒木玄さんの解説(X 2024年1月27日午後2:01 およびその後の補足)も役立つ。