Wilcoxon符号順位検定の対称性条件と変数変換
井口豊(生物科学研究所,長野県岡谷市)
最終更新: 2024 年 2 月 5 日
1. はじめに
Wilcoxon符号順位検定の適用条件に関しては,黒木玄さんの X (旧 Twitter) での解説が非常に重要である(X 2024年1月27日午後2:58)。
それに関連して,黒木さんの以下のポスト(X 2024年1月28日午後1:35)
超絶強い仮定は現実では成立していないと考えるべきなので、Mann-WhitneyのU検定やWilcoxonの符号順位検定の意味で「差がない」の意味は非常に分かりにくいです。
その辺の分かりにくさを認識せずにそれらの検定法を使っている人達はそれらの検定法を誤用している可能性が高いです。
これらのノンパラメトリック検定を安易に使わないほうが良い,ということである。
なお,対応あるデータに対する Brunner-Munzel 検定は,以下のページに記述した。
######################
黒木さんのポスト(X 2024年1月28日午後1:01),Mann-WhitneyのU検定やWilcoxonの順位検定に関するコンピュータシミュレーションでそれらを中央値に関する検定だと誤解して行うと失敗します
,を十分に念頭に置いた上で,以下の説明を読んで
ほしい。
ウィルコクソンの符号順位検定 (Wilcoxon signed-rank test) は,対応のある 2 群データに対するノンパラメトリック検定であり,中央値の検定として使われる。ただし,これは符号(付き)順位検定
の場合であり,独立 2 群に対するウィルコクソンの順位和検定 (Wilcoxon rank sum test),いわゆるマン-ホイットニー U 検定 (Mann-Whitney U test) は,中央値検定ではなく,順位平均検定なので,注意が必要である。
対応ある t 検定が,対応するデータの差を検定する 1 標本(1 群)検定(サンプル数 1 の検定)であることは,別ページに書いた。
独立 2 群の t 検定に対して,正規性や等分散性が仮定されるように, U 検定に対しても,等分散性が仮定される。
同様に,対応ある t 検定に対しては,差データの正規性が仮定され, Wilcoxon 符号順位検定に対しては,差データの分布対称性が仮定される。意外と,これらは忘れがちであり,特に,後者は知られていないのかもしれない。しかし,統計ソフト R の wilcox.test 関数のマニュアル Wilcoxon Rank Sum and Signed Rank Tests, Details には,以下のように書いてある。
a Wilcoxon signed rank test of the null that the distribution of x (in the one sample case) or of x - y (in the paired two sample case) is symmetric about mu is performed.
この説明は,ちょっと分かりづらいかもしれないが,要するに,差の分布が対称でない場合は,中央値の差ではなく,対称性からのズレを検出してしまうのである。
前述のように,実質的に 1 群検定なので,対応する 2 群が等分散かどうか,という問題は関係ないように見える。しかしながら,以下の例で示すように,対応する 2 群の分散が異なる場合に,その差データの分布が非対称となりがちである。
2. 差データ分布の非対称性問題とそれに対する分析手法
対数正規乱数データを使い,具体例を R の計算で見てみよう。中央値 5 は同じで,分散が異なる対数正規母集団からの標本の差のヒストグラムである。
#############
n<- 10000
set.seed(10)
x1 <- rlnorm(
n, meanlog = log(5), sdlog = 0.2
)
x2 <- rlnorm(
n, meanlog = log(5), sdlog = 0.8
)
hist(x2 - x1, freq = F, col = "royalblue")
################
結果は,次の図 1 のとおりである。
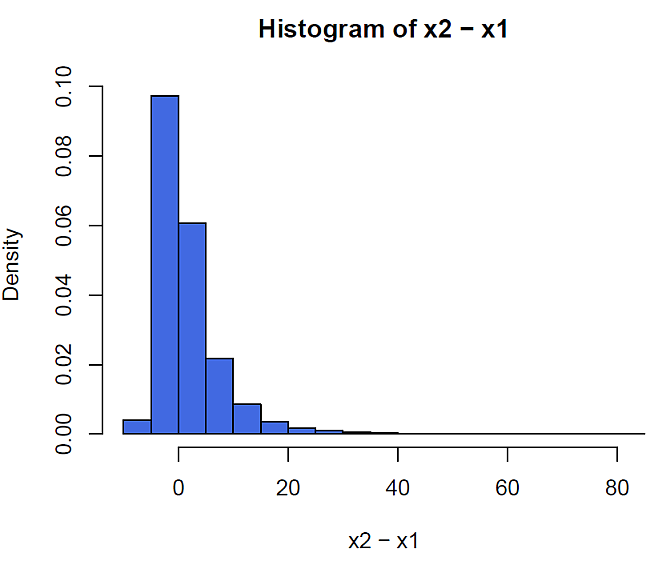
このように,差データに非対称が生じる場合の検定方法は,意外と単純である。変数変換をすれば良いのである。今の場合は,良く知られた対数変換である。
3. 非対称データと変換データの検定
前述と同じ設定で,大きさ 100 の対数正規乱数を 1 万回取り出し,生データと対数変換したデータで,それぞれ Wilcoxon 符号順位検定を実行して, p 値の分布を調べた。
#############
n<- 100
set.seed(10)
k<- 1e+4
p<- replicate(k, {
x1 <- rlnorm(
n, meanlog = log(5), sdlog = 0.2
)
log.x1<- log(x1)
x2 <- rlnorm(
n, meanlog = log(5), sdlog = 0.8
)
log.x2<- log(x2)
c(
wilcox.test(x1, x2,
correct = F, paired = TRUE
)$p.value,
wilcox.test(log.x1, log.x2,
correct = F, paired = TRUE
)$p.value
)
})
library(Hmisc)
par(mfrow = c(2, 2))
# Wilcoxon p 値出現頻度ヒストグラム
# 生データ
hist(
p[1, ],
xlab = "p value",
freq = F,
main = "Wilcoxon test for raw data"
)
# 対数変換データ
hist(
p[2, ],
xlab = "p value",
freq = F,
main = "Wilcoxon test for log data"
)
# 経験累積分布関数(ECDF)
ck<- 1:k/k
d.1<- c(ck, p[1, ])
d.2<- c(ck, p[2, ])
test<- relevel(factor(
rep(c("Theoretical", "Wilcoxon"), each = k)
), ref = "Theoretical")
# 生データ
Ecdf(
d.1,
xlab="p value",
label.curves=list(keys= 1:2),
lty = 1:2,
group = test,
main = "ECDF"
)
# 対数変換データ
Ecdf(
d.2,
xlab="p value",
label.curves=list(keys= 1:2),
lty = 1:2,
group = test,
main = "ECDF"
)
################
結果は,次の図 2 のとおりである。
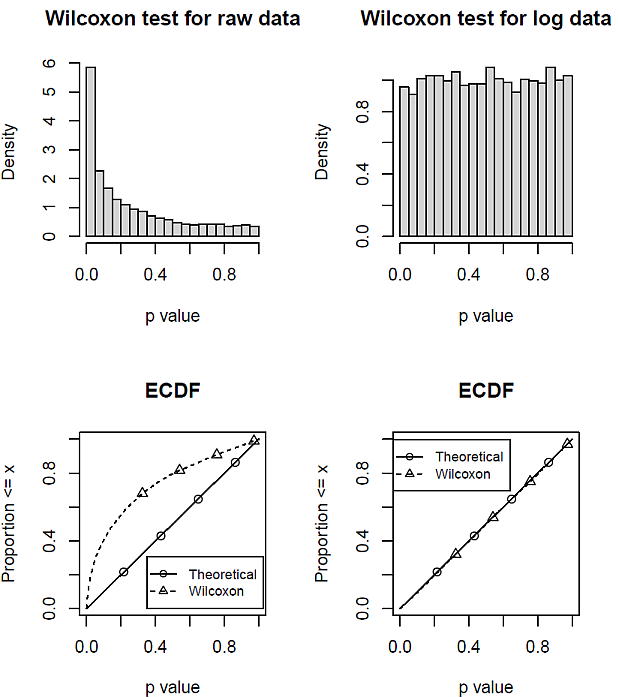
差データが非対称な分布の場合は,そのまま Wilcoxon 符号順位検定すると, p 値が片寄って出現してしまうのである。
ただし,ここで黒木さんの以下のコメント(X 2024年1月27日午後2:01)参照。
これに従うと,そもそも,上記のようなWilcoxonの符号順位和検定の「中央値を中心に対称」という条件からの逸脱について調べるときには、「中央値は0」ではなく、「P(X₁+X₂>0)=1/2」という条件の下でのシミュレーションを行うべきである。
中央値が等しいという単純な条件で,この検定を使うこと自体に問題あり,なのである。
一方で,対数変換して,対称な分布に直すと,一様に p 値が現れる。実は,対数正規分布データを対数変換したので,正規分布データとなり,パラメトリックな対応ある t 検定が適用できる状態なのだ。
単純な対数変換だけでなく, Box-Cox 変換や Yeo-Johnson 変換もある。正規分布でないからと,安易にノンパラメトリック検定にとびつくのではなく,まずデータ分布の特徴をよく見て,変数変換や一般化線形モデルの利用も探ってみるべきである。
X (旧 Twitter) で,黒木玄さんが, Kullback-Leibler 情報量 (Kullback-Leibler Divergence) に言及していた(X 2024年1月19日午後0:30)。いつもながら有益な情報を与えてくれる。その解説書, Kullback-Leibler情報量とSanovの定理,最後のページの説明, Kullback-Leibler 情報量には「Shannon 情報量からの平均符号長の増分」という解釈が可能である
,ということである。 R のパッケージ, philentropy の関数,distance の Details を見ると, Kullback-Leibler が Shannon's entropy family に分類されていることが分かる。
生態学研究者にとっては,情報量と言うと, Shannon 関数,あるいは, Shannon-Weaver 関数のほうが馴染み深いような気がする。黒木玄さんの解説の中で, Stirling の公式が出てきたので,ふと思い出して,学生時代に学んだ教科書,動物群集研究法I-多様性と種類組成-
(木元 新作,1976)を見返し, Stirling の公式を用いて, Shannon-Weaver 関数を導くことが説明されていて(p.61-64),懐かしくなった。
このパッケージにある KL 関数を使って, Kullback-Leibler 情報量を求めて,ヒートマップ図で,前述の理論値,生データ,対数変換データを比べて見た。
#############
# Kullback-Leibler 情報量
library(philentropy)
KLmat<- KL(
rbind(
ck/sum(ck),
p[1, ]/sum(p[1, ]),
p[2, ]/sum(p[2, ])
), unit = "log")
dimnames(KLmat)<- list(
c("Theor", "Raw", "Log"),
c("Theor", "Raw", "Log")
)
KLmat
# ヒートマップ
heatmap(
KLmat,
cexRow = 1.5,
cexCol = 1.5
)
################
Kullback-Leibler 情報量の結果は,以下のように出力される。
Theor Raw Log
Theor 0.0000000 1.1559778 0.4923631
Raw 1.1559778 0.0000000 0.9913163
Log 0.4923631 0.9913163 0.0000000
ヒートマップは,次の図 3 のとおりである。
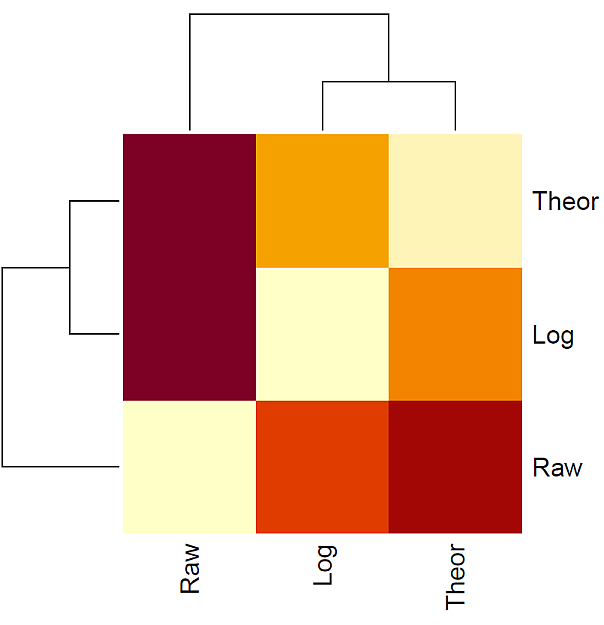
対数変換データの計算結果のほうが,理論値に近くなっている。